Метилтрансфераза
Метилтрансферази — це велика група ферментів, які метилюють свої субстрати.

Їх можна розділити на кілька підкласів на основі їх структурних особливостей. Найпоширенішим класом метилтрансфераз є клас I, усі вони містять складку Россмана[en] для зв'язування S-аденозилметіоніну (англ. SAM). Метилтрансферази класу II містять домен SET, прикладом якого є гістонові метилтрансферази[en] домену SET, і метилтрансферази класу III, які пов'язані з мембраною.[1] Метилтрансферази також можна згрупувати як різні типи, що використовують різні субстрати в реакціях перенесення метилу. Ці типи включають білкові метилтрансферази, ДНК/РНК метилтрансферази, метилтрансферази природних продуктів і незалежні від SAM метилтрансферази. SAM є класичним донором метилу для метилтрансфераз, однак у природі зустрічаються приклади інших донорів метилу. Загальним механізмом перенесення метилу є SN2[en]-подібна нуклеофільна атака, де сірка метіоніну служить групою, що відходить, а приєднана до неї метильна група діє як електрофіл, який переносить метильну групу на субстрат ферменту. Під час цього процесу SAM перетворюється на S-аденозилгомоцистеїн (англ. SAH). Розрив зв'язку SAM-метил і утворення зв'язку субстрат-метил відбуваються майже одночасно. Ці ферментативні реакції зустрічаються в багатьох шляхах і причетні до генетичних захворювань, раку та метаболічних захворювань. Іншим типом переносу метилу є радикал S-аденозилметіонін, який є метилюванням неактивованих атомів вуглецю в первинних метаболітах, білках, ліпідах і РНК.
Функція ред.
Генетика ред.
Метилювання, як і інші епігенетичні модифікації, впливає на транскрипцію, стабільність генів і батьківський імпринтинг.[2] Він безпосередньо впливає на структуру хроматину та може модулювати транскрипцію генів або навіть повністю заглушити чи активувати гени без мутації самого гена. Хоча механізми цього генетичного контролю є складними, гіпо- та гіпер- метилювання ДНК причетне до багатьох захворювань.
Білкова регуляція ред.
Метилювання білків відіграє регулювальну роль у взаємодіях білок-білок, взаємодіях білок-ДНК та активації білка.
Приклади: RCC1[3], важливий мітотичний білок, метилюється, щоб він міг взаємодіяти з центромерами хромосом. Це приклад регуляції білок-білкової взаємодії, оскільки метилювання регулює приєднання RCC1 до гістонових білків H2A та H2B. Взаємодія RCC1-хроматин також є прикладом взаємодії білок-ДНК, оскільки інший домен RCC1 взаємодіє безпосередньо з ДНК, коли цей білок метилюється. Коли RCC1 не метильований, клітини, що діляться, мають кілька полюсів веретена і зазвичай не можуть вижити.
p53 метилюється на лізині для регулювання його активації та взаємодії з іншими білками у відповідь на пошкодження ДНК. Це приклад регуляції білок-білкової взаємодії та активації білка. p53 є відомим супресором пухлин[en], який активує шляхи відновлення ДНК, ініціює апоптоз і призупиняє клітинний цикл. Загалом він реагує на мутації в ДНК, сигналізуючи клітині виправити їх або ініціювати загибель клітини, щоб ці мутації не сприяли раку.
NF-κB[en] (білок, що бере участь у запаленні) є відомою мішенню метилювання метилтрансферазиSETD6, яка вимикає передачу сигналів NF-κB шляхом інгібування однієї з її субодиниць, RelA. Це зменшує активацію транскрипції та запальну відповідь, роблячи метилювання NF-κB регуляторним процесом, за допомогою якого зменшується передача клітинних сигналів через цей шлях.[4]
Природні продукти метилтрансферази забезпечують різноманітні вхідні дані в метаболічні шляхи, включаючи доступність кофакторів, сигнальних молекул і метаболітів. Це регулює різні клітинні шляхи, контролюючи активність білка.
Типи ред.
Гістонові метилтрансферази ред.
Гістонметилтрансферази мають вирішальне значення для генетичної регуляції на епігенетичному рівні. Вони модифікують головним чином лізин на ε-азоті та аргінін гуанідінієву групу на хвостах гістонів. Лізинметилтрансферази та аргінінметилтрансферази є унікальними класами ферментів, але обидва вони зв'язують SAM як донор метилу для своїх субстратів гістонів. Амінокислоти лізину можуть бути модифіковані однією, двома або трьома метильними групами, тоді як амінокислоти аргініну можуть бути модифіковані однією або двома метильними групами. Це збільшує силу позитивного заряду та гідрофобність залишків, що дозволяє іншим білкам розпізнавати метильні мітки. Ефект цієї модифікації залежить від розташування модифікації на хвості гістону та інших модифікацій гістону навколо нього. Розташування модифікацій може частково визначатися послідовністю ДНК, а також малими некодувальними РНК і метилюванням самої ДНК. Найчастіше це гістони H3 або H4, які метилюються у хребетних. Навколо модифікації може відбутися або збільшення, або зниження транскрипції генів. Збільшення транскрипції є результатом зниження конденсації хроматину, тоді як зниження транскрипції є результатом збільшення конденсації хроматину.[5] Метильні позначки на гістонах сприяють цим змінам, слугуючи місцями для залучення інших білків, які можуть додатково модифікувати хроматин.[6]
N-кінцеві метилтрансферази ред.
N-альфа-метилтрансферази переносять метильну групу від SAM до N-кінцевого азоту на білкових мішенях. N-кінцевий метіонін спочатку розщеплюється іншим ферментом, а консенсусна послідовність X-Пролін-Лізин розпізнається метилтрансферазою. Для всіх відомих субстратів амінокислотою X є аланін, серин або пролін. Ця реакція дає метильований білок і SAH. Відомі мішені цих метилтрансфераз у людей включають RCC-1 (регулятор ядерних транспортних білків) і білок ретинобластоми (білок-супресор пухлин, який пригнічує надмірний поділ клітин). Метилювання RCC-1 є особливо важливим у мітозі, оскільки воно координує локалізацію деяких ядерних білків за відсутності ядерної оболонки. Коли RCC-1 не метильований, поділ клітин відбувається ненормально після утворення додаткових полюсів веретена.[7] Функція N-кінцевого метилювання білка ретинобластоми невідома.
ДНК/РНК метилтрансферази ред.
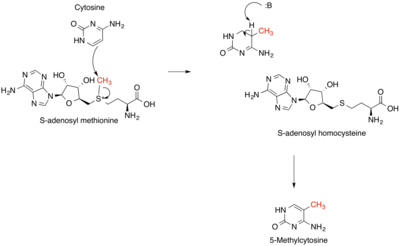
Метилювання ДНК, ключовий компонент генетичної регуляції, відбувається головним чином на 5-вуглецевому атомі основи цитозину, утворюючи 5'-метилцитозин.[8] Метилювання — це епігенетична модифікація, що каталізується ферментами ДНК-метилтрансферази, включаючи DNMT1, DNMT2 (перейменований на TRDMT1, щоб відобразити його функцію метилювання тРНК, а не ДНК) і DNMT3. Ці феноми використовують S-аденозилметіонін як донор метилу та містять кілька висококонсервативних структурних особливостей між трьома формами; вони включають сайт зв'язування S-аденозилметіоніну, сусідню пролін-цистеїнову пару, яка утворює тіолат-аніон, важливий для механізму реакції, і кишеню зв'язування субстрату цитозину. Багато властивостей ДНК-метилтрансфераз «високо» зберігаються впродовж багатьох класів життя, від бактерій до ссавців. Окрім контролю експресії певних генів, існують різноманітні білкові комплекси, багато з яких впливають на здоров'я людини, які зв'язуються лише з метильованими сайтами розпізнавання ДНК. Вважається, що багато ранніх ДНК-метилтрансфераз були похідним від РНК-метилтрансфераз, які малибути активними у світі РНК для захисту багатьох видів примітивних РНК.[9] Метилювання РНК спостерігалося в різних типах видів РНК, а саме: мРНК, рРНК, тРНК, сноРНК, снРНК, мікроРНК, тмРНК, а також вірусні види РНК. Специфічні РНК-метилтрансферази використовуються клітинами для позначення їх на видах РНК відповідно і до потреб середовища, що панує навколо клітин, що є частиною галузі, яка називається молекулярною епігенетикою. 2'-O-метилювання, метилювання m6A, метилювання m1G, а також m5C є найпоширенішими ознаками метилювання, які спостерігаються в різних типах РНК.
6A — це фермент, який каталізує таку хімічну реакцію:[10]
S-аденозил-L-метіонін + ДНК аденін S-аденозил-L-гомоцистеїн + ДНК 6-метиламінопурин
m6A в основному знаходили в прокаріотах до 2015 року, коли його також ідентифікували в деяких еукаріотів. Метилтрансферази m6A метилюють аміногрупу в ДНК у положенні C-6 спеціально, щоб запобігти системі хазяїна перетравлювати власний геном за допомогою ферментів рестрикції.[11]
m5C відіграє роль у регуляції транскрипції генів. Трансферази m5C є ферментами, які виробляють C5-метилцитозин у ДНК у положенні C-5 цитозину та зустрічаються в більшості рослин і деяких еукаріотів.[12]
Натуральний продукт метилтрансферази ред.
Метилтрансферази природного продукту (NPMTs) — це різноманітна група ферментів, які додають метильні групи до природних малих молекул. Як і багато інших метилтрансфераз, SAM використовується як донор метилу, і утворюється SAH. Метильні групи додаються до атомів S, N, O або C і класифікуються за тим, які з цих атомів модифіковані, причому O-метилтрансферази представляють найбільший клас. Метильовані продукти цих реакцій виконують різноманітні функції, включно з кофакторами, пігментами, сигнальними сполуками та метаболітами. NPMTs можуть виконувати регуляторну роль, змінюючи реактивність і доступність цих сполук. Ці ферменти не дуже консервативні для різних видів, оскільки вони виконують більш специфічну функцію, забезпечуючи невеликі молекули для спеціалізованих шляхів у видів або менших груп видів. Відображенням цього розмаїття є різноманітність каталітичних стратегій, включаючи загальний кислотно-основний каталіз, каталіз на основі металів[13], а також ефекти близькості та десольватації, які не вимагають каталітичних амінокислот. NPMT є найбільш функціонально різноманітним класом метилтрансфераз.[14]
Важливими прикладами цього класу ферментів у людини є фенілетаноламін-N-метилтрансфераза (PNMT), яка перетворює норадреналін в адреналін,[16] і гістамін-N-метилтрансфераза (HNMT), яка метилює гістамін у процесі метаболізму гістаміну.[17] Катехол-O-метилтрансфераза (COMT) руйнує клас молекул, відомий як кахоламіни, який включає дофамін, епінефрин і норепінеферин.[18]
Незалежні від SAM метилтрансферази ред.
Метанол, метилтетрагідрофолат, моно-, ди- і триметиламін, метанетіол, метилтетрагідрометаноптерин і хлорметан є донорами метилу, які зустрічаються в біології як донори метильної групи, як правило, у ферментативних реакціях з використанням кофактора вітаміну B12.[19] Ці субстрати беруть участь у шляхах перенесення метилу, включаючи біосинтез метіоніну, метаногенез та ацетогенез.
Радикальні SAM метилтрансферази ред.
Виходячи з різних білкових структур і механізмів каталізу, існують 3 різні типи радикальних SAM (RS) метилаз: клас A, B і C. RS метилази класу A найкраще охарактеризовані з 4 ферментів і пов'язані як з RlmN, так і з RlmN. Cfr. RlmN є всюдисущим у бактеріях, що підвищує точність трансляції, а RlmN каталізує метилювання C2 аденозину 2503 (A2503) у 23 S рРНК і C2 аденозину (A37). Cfr, з іншого боку, також каталізує метилювання C8 з A2503, а також каталізує метилювання C2.[20] Клас B наразі є найбільшим класом радикальних SAM метилаз, які можуть метилювати як sp2-гібридизовані, так і sp3-гібридизовані атоми вуглецю в різних наборах субстратів, на відміну від класу A, який каталізує лише sp2-гібридизовані атоми вуглецю. Основною відміністю, яка відрізняє клас B від інших, є додатковий N-кінцевий домен зв'язування кобаламіну, який зв'язується з доменом RS.[21] Метилаза класу C має гомологічну послідовність із ферментом RS, копропорфіриноген III оксидазою (HemN), який також каталізує метилювання sp2-гібридизованих вуглецевих центрів, але в ній відсутні 2 цистеїни, необхідні для метилювання в механізмі класу A.[20]
Клінічне значення ред.
Як і будь-який біологічний процес, який регулює експресію та/або функцію генів, аномальне метилювання ДНК пов'язане з генетичними розладами, такими як ICF, синдром Ретта та синдром крихкої Х-хромосоми.[2] Ракові клітини зазвичай виявляють меншу активність метилювання ДНК загалом, хоча часто гіперметилювання в місцях, які не метильовані в нормальних клітинах; це надмірне метилювання часто функціонує як спосіб інактивації генів-супресорів пухлин. Пригнічення загальної активності ДНК-метилтрансферази запропонували як варіант лікування, але інгібітори DNMT, аналоги їх субстратів цитозину, виявилися високотоксичними через їх схожість з цитозином; ця подібність до нуклеотиду спричиняє включення інгібітора в трансляцію ДНК, що призводить до синтезу ДНК що не функціонує.
Метилаза, яка змінює сайт зв'язування рибосомальної РНК антибіотика лінезоліду, викликає перехресну резистентність до інших антибіотиків, які діють на рибосомну РНК. Плазмідні вектори, здатні передавати цей ген, є причиною потенційно небезпечної перехресної резистентності.[22]
Приклади ферментів метилтрансфераз, що стосуються захворювання:
- тіопуринметилтрансфераза: дефекти в цьому гені спричиняють токсичне накопичення тіопуринових сполук, препаратів, що використовуються в хіміотерапії та імуносупресивній терапії
- метіонінсинтаза: злоякісна анемія, викликана дефіцитом вітаміну В12, спричинена відсутністю кофактора для ферменту метіонінсинтази
Застосування у відкритті та розробці ліків ред.
Недавня робота показала, що метилтрансферази беруть участь у метилюванні природних протипухлинних агентів для використання аналогів S-аденозилметіоніну (SAM), які несуть альтернативні алкільні групи як заміну метилу. Розробка легкої хемоферментної платформи для генерування та використання диференціально алкілованих аналогів SAM у контексті відкриття[en] та розробляння ліків відома як алкілрандомізація.[23]
Застосування в лікуванні раку ред.
У клітинах людини виявлено, що m5C асоціюється з аномальними пухлинними клітинами при раку.[24] Роль і потенційне застосування m5C включає в себе балансування порушеної ДНК при раку як гіперметилювання, так і гіпометилювання. Епігенетичне відновлення ДНК може бути застосоване шляхом зміни кількості m5C в обох типах ракових клітин (гіперметилювання/гіпометилювання), а також середовища раку для досягнення еквівалентної точки для інгібування пухлинних клітин.[25]
Приклади ред.
- Катехол-О-метилтрансфераза
- ДНК-метилтрансфераза
- Гістон метилтрансфераза
- 5-Метилтетрагідрофолат-гомоцистеїн метилтрансфераза
- О-метилтрансфераза
- метіонінсинтази
- кориноїдно-залізо-сірчаний білок
Примітки ред.
- ↑ Katz, J. E.; Dlakic, M; Clarke S (18 липня 2003). Automated identification of putative methyltransferases from genomic open reading frames. Molecular & Cellular Proteomics. 2 (8): 525—40. doi:10.1074/mcp.M300037-MCP200. PMID 12872006.
- ↑ а б Siedlecki, P; Zielenkiewicz, P (2006). Mammalian DNA methyltransferases. Acta Biochimica Polonica. 53 (2): 245—56. doi:10.18388/abp.2006_3337. PMID 16582985.
- ↑ RCC1(human) PhosphoSite
- ↑ Levy, Dan та ін. (5 грудня 2010). Lysine methylation of the NF-κB subunit RelA by SETD6 couples activity of the histone methyltransferase GLP at chromatin to tonic repression of NF-κB signaling. Nature Immunology. 12 (1): 29—36. doi:10.1038/ni.1968. PMC 3074206. PMID 21131967.
- ↑ Turner, Bryan M. (2001). Chromatin and gene regulation : mechanisms in epigenetics. Malden, MA: Blackwell Science. ISBN 978-0865427433.
- ↑ Greer, Eric L.; Shi, Yang (3 квітня 2012). Histone methylation: a dynamic mark in health, disease and inheritance. Nature Reviews Genetics. 13 (5): 343—357. doi:10.1038/nrg3173. PMC 4073795. PMID 22473383.
- ↑ Clarke, Paul (May 2007). Anchoring RCC1 by the tail. Nature Cell Biology. 9 (5): 485—487. doi:10.1038/ncb0507-485. PMID 17473856.
- ↑ Lan, J; Hua, S; He, X; Zhang, Y (2010). DNA methyltransferases and methyl-binding proteins of mammals. Acta Biochimica et Biophysica Sinica. 42 (4): 243—52. doi:10.1093/abbs/gmq015. PMID 20383462.
- ↑ Rana, Ajay K.; Ankri, Serge (1 січня 2016). Reviving the RNA World: An Insight into the Appearance of RNA Methyltransferases. Frontiers in Genetics. 7: 99. doi:10.3389/fgene.2016.00099. PMC 4893491. PMID 27375676.
- ↑ Kessler, Christoph; Manta, Vicentiu (1 січня 1990). Specificity of restriction endonucleases and DNA modification methyltransferases — a review (edition 3). Gene (англ.). 92 (1): 1—240. doi:10.1016/0378-1119(90)90486-B. ISSN 0378-1119. PMID 2172084.
- ↑ Narva, Kenneth E.; Van Etten, James L.; Slatko, Barton E.; Benner, Jack S. (25 грудня 1988). The amino acid sequence of the eukaryotic DNA [N6-adenine]methyltransferase M·CviBIII, has regions of similarity with the prokaryotic isoschizomer M · TaqI and other DNA [N6-adenine] methyltransferases. Gene (англ.). 74 (1): 253—259. doi:10.1016/0378-1119(88)90298-3. ISSN 0378-1119. PMID 3248728.
- ↑ Posfai, Janos; Bhagwat, Ashok S.; Roberts, Richard J. (25 грудня 1988). Sequence motifs specific for cytosine methyltransferases. Gene (англ.). 74 (1): 261—265. doi:10.1016/0378-1119(88)90299-5. ISSN 0378-1119. PMID 3248729.
- ↑ A2. Metal Ion or Electrostatic Catalysis LibreTexts
- ↑ Liscombe, David K.; Louie, Gordon V.; Noel, Joseph P. (2012). Architectures, mechanisms and molecular evolution of natural product methyltransferases. Natural Product Reports. 29 (10): 1238—50. doi:10.1039/c2np20029e. PMID 22850796.
- ↑ Ashihara, Hiroshi; Yokota, Takao; Crozier, Alan (2013). Biosynthesis and catabolism of purine alkaloids. Advances in Botanical Research. Т. 68. с. 111—138. doi:10.1016/B978-0-12-408061-4.00004-3. ISBN 9780124080614.
- ↑ PNMT phenylethanolamine N-methyltransferase. NCBI Genetic Testing Registry. Процитовано 18 лютого 2014.
- ↑ HNMT histamine N-methyltransferase. NCBI Genetic Testing Registry. Процитовано 18 лютого 2014.
- ↑ COMT catechol-O-methyltransferase. NCBI Genetic Testing Registry. Процитовано 18 лютого 2014.
- ↑ Ragsdale, S.W. «Catalysis of methyl group transfers involving tetrahydrofolate and B12» Vitamins and Hormones, 2008.
- ↑ а б Bauerle, Matthew R.; Schwalm, Erica L.; Booker, Squire J. (13 лютого 2015). Mechanistic Diversity of Radical S-Adenosylmethionine (SAM)-dependent Methylation. The Journal of Biological Chemistry. 290 (7): 3995—4002. doi:10.1074/jbc.R114.607044. ISSN 0021-9258. PMC 4326810. PMID 25477520.
- ↑ Sofia, H. J.; Chen, G.; Hetzler, B. G.; Reyes-Spindola, J. F.; Miller, N. E. (1 березня 2001). Radical SAM, a novel protein superfamily linking unresolved steps in familiar biosynthetic pathways with radical mechanisms: functional characterization using new analysis and information visualization methods. Nucleic Acids Research. 29 (5): 1097—1106. doi:10.1093/nar/29.5.1097. ISSN 1362-4962. PMC 29726. PMID 11222759.
- ↑ Morales G, Picazo JJ, Baos E, Candel FJ, Arribi A, Peláez B, Andrade R, de la Torre MA, Fereres J, Sánchez-García M (March 2010). Resistance to linezolid is mediated by the cfr gene in the first report of an outbreak of linezolid-resistant Staphylococcus aureus. Clin. Infect. Dis. 50 (6): 821—5. doi:10.1086/650574. PMID 20144045.
- ↑ Singh, S; Zhang, J; Huber, TD; Sunkara, M; Hurley, K; Goff, RD; Wang, G; Zhang, W; Liu, C (7 квітня 2014). Facile chemoenzymatic strategies for the synthesis and utilization of S-adenosyl-(L)-methionine analogues. Angewandte Chemie International Edition in English. 53 (15): 3965—9. doi:10.1002/anie.201308272. PMC 4076696. PMID 24616228.
- ↑ Jones, Peter A. (1 червня 1996). DNA Methylation Errors and Cancer. Cancer Research (англ.). 56 (11): 2463—2467. ISSN 0008-5472. PMID 8653676.
- ↑ D, Hanahan; Ra, Weinberg (4 березня 2011). Hallmarks of Cancer: The Next Generation. Cell (англ.). 144 (5): 646—74. doi:10.1016/j.cell.2011.02.013. PMID 21376230.
Посилання ред.
- MeSH Methyltransferases
- 3-D Structure of DNA Methyltransferase
- A novel methyltransferase: the 7SK snRNA Methylphosphate Capping Enzyme as seen on Flintbox
- «The Role of Methylation in Gene Expression» on Nature Scitable
- «Nutrition and Depression: Nutrition, Methylation, and Depression» on Psychology Today
- «DNA Methylation — What is DNA Methylation?» from News-Medical.net
- «Histone Lysine Methylation» Genetic pathways involving Histone Methyltransferases from Cell Signaling Technology